Pour manipuler les données, le SQL est très pratique. Mais cela demande d’ecrire les actions à réaliser en 1 seul bloc. Avec R, c’est beaucoup plus facile, car il est possible de décomposer les actions une à une, les unes à la suite des autres (à l’aide de la librairie dplyr). Cet article va lister les équivalences entre R et SQL pour chaque action à réaliser.
Pour illustrer la manière d’ecrire les actions en SQL et en R, on va utiliser la table de données suivante :
| Personne | Matiere | Note |
| A | Maths | 13 |
| B | Maths | 11 |
| A | Anglais | 15 |
| B | Anglais | 12 |
Pour sélectionner les colonnes pour lesquels on souhaite récupérer les données :
SQL :
SELECT Note
FROM maTable
R :
library(dplyr)
maTable %>% select(Note)
Pour filtrer les données :
SQL :
SELECT Note
FROM maTable
WHERE Matiere = "Maths"
R:
library(dplyr)
maTable %>% filter(Matiere == "Maths) %>% select(Note)
Pour faire un décompte avec regroupement :
SQL :
SELECT Matiere, AVG(Note) as Moyenne
FROM maTable
GROUP BY Matiere
R :
library(dplyr)
maTable %>% group_by(Matiere) %>% summarise(Moyenne=mean(Note))
Pour faire un tri :
SQL :
SELECT Note
FROM maTable
ORDER BY Note DESC
R :
library(dplyr)
maTable %>% select(Note) %>% arrange(desc(Note))
Pour faire une union de 2 tables de données :
SQL :
(SELECT *
FROM maTable)
UNION
(SELECT *
FROM maTable2)
R :
library(dplyr)
maTable %>% bind_rows(maTable2)
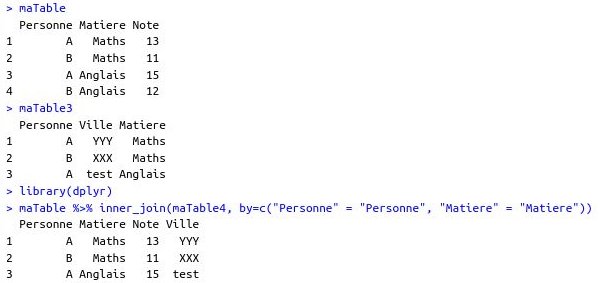
Pour faire une jointure de 2 tables de données :
SQL :
SELECT *
FROM maTable
INNER JOIN maTable3 ON maTable.Personne = maTable3.Personne AND maTable.Matiere = maTable3.Matiere
R :
library(dplyr)
maTable %>% inner_join(maTable3, by=c("Personne" = "Personne", "Matiere" = "Matiere"))
Pour + d’infos :
https://statisticsglobe.com/r-dplyr-join-inner-left-right-full-semi-anti